GSEA
Install
- GSEAのサイトにログイン
- Windows用のGSEAをダウンロード/インストール
Dataの準備 (RNA-seq)
- HISAT2-Stringtie-PrepDE.py3のアウトプットgene_matrix.csvなどcountデータを準備する. (TPMデータなどの補正データは使用できない.)
- gene_matrix.csvをDESeq2を用いてNormalized Countsデータに変換する.
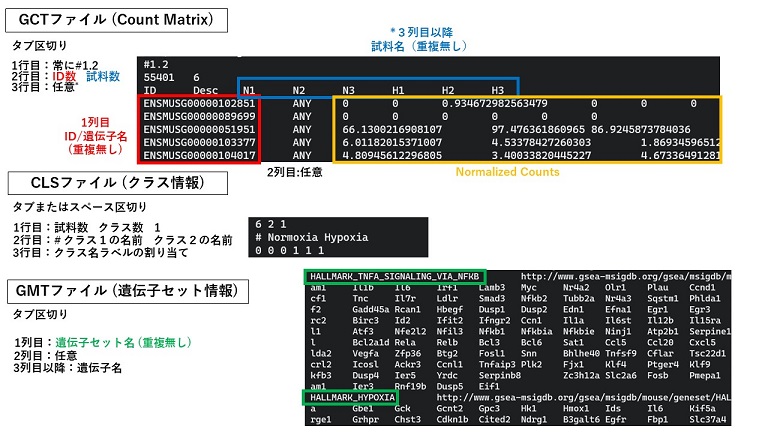
- 以下を参考に*.gct, *.clsを作る.*.gmtは作成またはGSEAサイトなどからダウンロードする.
参考用データ:
GCTファイル,
CLSファイル,
GMTファイル
解析の実行
- GSEAを起動する.
- Load data
- *.gct, *.clsをアップロード. 必要なら*.gmtも.
- Run GSEA
- Expression dataset: *.gct
- Gene set database: 適当なものを選択
- Number of Permutations: 1000
- Phenotype labels: *.cls (Treat_versus_Control)
- Collapse/Remap to gene symbols: Collapse (遺伝子名でなくIDを使用している場合)
- Permutation Type: gene_set
- Chip Platform: IDに合わせて適当なものを選択
- Run
- GSEA Reports (C:\Users\username\gsea_home\output\配下に結果格納されている.)